 https://docs.scipy.org/doc/numpy/reference/index.html
https://docs.scipy.org/doc/numpy/reference/index.html(rev. 2020-07-10)
Ce support de cours a pour vocation de faire une introduction rapide de la librairie Numpy en couvrant les fonctions essentielles à son utilisation, en particulier la manipulation d’objets ndarray. Il n’a pas pour but d’être exhaustif, ni dans le choix des fonctions, ni dans les paramètres des fonctions documentées.
Les informations originales et complètes peuvent être trouvées en suivant les lien qui accompagnent chaque chapitre.
Pour accéder à la documentation complète de NumPy, vous pouvez vous rendre sur  https://docs.scipy.org/doc/numpy/reference/index.html
https://docs.scipy.org/doc/numpy/reference/index.html
Le Jupyter Notebook d’exercices associé à ce cours peut être téléchargé ici.
Historiquement, le calcul scientifique avait pour language de programmation clé Fortran. Ensuite sont venus C, C++, Matlab/Octave. Plus récemment, d’autres langages ont rejoint ce cercle. On y trouve Python, grace en particulier à la librairie Numpy, mais aussi les langages R et Julia.
Pour tendre vers de meilleures performances en calcul, en plus de la parallélisation du code, l’ingénieur va s’intéresser à la quantité de mémoire réservée et le temps de calcul nécessaire pour faire une tâche. Plus le langage est de bas niveau, plus l’utilisation de la mémoire et du processeur pourront être efficaces.
Selon ce critère, l’assembleur serait le meilleur langage puisque il est au plus bas niveau possible. Dans les faits, il n’est pas courament utilisé pour ça étant donné qu’il requiert que l’ingénieur décompose chaque opération en série d’instructions processeur. Cela finit par des quantités de code colossales et peu lisibles, même pour réaliser des tâches simples. On peut ajouter à cela le fait qu’il n’est pas portable d’un type de processeur à un autre.
Viennent ensuite Fortran, C, C++ qui sont des langages de niveau intermédiaire. Ils doivent être compilés avant de pouvoir être exécutés par le processeur. Ces langages permettent une grande optimisation des ressources. C’est leur grande force. Leurs inconvénients sont le temps et la quantité de code nécessaires à réaliser des tâches simples ainsi que la nécessité de gérer l’espace mémoire manuellement.
Finalement viennent les langages de haut niveau, tel que Matlab/Octave, Python, R. Il s’agit de langages dits “interprétés” parce qu’ils sont convertis à la volée en un code intermédiaire et exécutés par une application “interprète”. Ceux-ci offrent des avantages considérables pour le développeur, notamment la gestion automatisée de la mémoire, une syntaxe plus haut-niveau (et donc plus proche de l’humain). Ces avantages ont un coût … à savoir que la gestion de la mémoire est moins optimisée (se faisant automatiquement). Le temps de calcul est également impacté par des traitements automatiques supplémentaires que le langage doit ajouter pour compenser les facilités offertes au développeur.
Avec un langage de haut niveau, il est clair que le temps de développement sera plus court et le temps d’exécution sera plus long qu’avec un langage de niveau plus bas. Pour cette raison il est important de prendre le temps d’évaluer et chercher le bon rapport entre coût et gain lors du choix de la technologie employée pour un projet.
Une approche qui est souvent employée consiste à coder la grande partie du projet dans un langage de haut niveau et écrire les portions cruciales du code dans un langage de plus bas niveau. Ceci permet de trouver un bon compromis, alliant les avantages de chaque language. Une autre approche largement employée et proche de la précédente consiste à utiliser un langage de haut niveau et d’employer une librairie qui optimise les ressources employées pour faire les calculs coûteux. L’utilisation de la librairie Numpy nous fait entrer dans ce cas de figure.
L’analyse du temps d’exécution à l’aide d’un profiler permet de localiser ces parties critiques.
NumPy est la librairie qui offre la pièce fondamentale pour faire du calcul scientifique avec Python. Elle est sous licence BSD, ce qui permet à chacun de l’utiliser librement pour ses besoins (académiques, privés et professionnels).
Elle est majoritairement écrite en C (pour les parties clé à optimiser) et Python. Quelques portions sont également écrites en C++ et Fortran.
Elle étend les capacités de Python pour travailler sur des tableaux et matrices à n dimensions de façon bien plus optimisée et offre des fonctions mathématiques de haut niveau sur ces objets.
Pour les utilisateurs connaissant déjà l’environnement Matlab / Octave, il sera d’une grande aide de consulter cette page https://docs.scipy.org/doc/numpy/user/numpy-for-matlab-users.html qui relate les principales différences et équivalences entre ceux-ci et Numpy.
Par convention, la communauté importe Numpy de la façon suivante. Ceci permet de faire appel à des fonctions avec la notation plus courte : np.array() (plutôt que numpy.array()).
import numpy as npL’objet ndarray pour N-dimensional array est l’élément central de la librairie Numpy. Tout ce qui est décrit ci-dessous a pour vocation de travailler sur ces objets, de la création, aux opérations en passant par leurs attributs et les manipulations possibles.
 Dans cette documentation, il est mention d’objets de type ndarray et d’autres de type array. Voici la différence :
Dans cette documentation, il est mention d’objets de type ndarray et d’autres de type array. Voici la différence :
np.array.Voici encore d’autres termes qui seront régulièrement employés :
On peut se demander l’intérêt d’utiliser une librairie spécifique pour travailler avec des listes/matrices/arrays alors que Python fourni déjà les objets de type list() écrit plus rapidement [ ], ainsi qu’une série de fonctions qui leurs sont utiles. Une rapide comparaison est proposée en fin de ce document au chapitre Comparaison entre une liste Python et un Array Numpy.
np.array(object, dtype=None, copy=True, order='K',
subok=False, ndmin=0)Le tableau à N-dimensions est l’élément central de la librairie Numpy. Il peut être créé de plusieurs façons. La première est en utilisant np.array().
object un array ou objet exposant une interface de type array. Il peut être à n-dimensions.
dtype est le type choisi pour les données qui y seront stockées.
Voici les principaux types disponibles :
np.bool : Booléen (True|False)np.int8 : Entier (-128 à 127)np.int16 : Entier (-2¹⁵ à 2¹⁵-1)np.int32 : Entier (-2³¹ à 2³¹-1)np.int64 : Entier (-2⁶³ à 2⁶³-1) (défaut pour les entiers)np.uint8 : Entier (0 à 255)np.uint16 : Entier (0 à 2¹⁶-1)np.uint32 : Entier (0 à 2³²-1)np.uint64 : Entier (0 à 2⁶⁴-1)np.float16 : Flottant (demi-précision flottant)np.float32 : Flottant (simple-précision flottant)np.float64 : Flottant (double-précision flottant) (défaut pour les flottants)np.complex64 : Complexe (simple-précision flottant sur les 2 valeurs)np.complex128 : Complexe (double-précision flottant sur les 2 valeurs)a = np.array([6.1, 7.2, 8.3])
a
# array([6.1, 7.2, 8.3])
type(a)
# numpy.ndarray
a.dtype
# dtype('float64')
b = np.array([1, 2, 3], dtype=np.uint64)
b
# array([1, 2, 3], dtype=uint64)
b.dtype
# dtype('uint64')
np.array([[1, 2, 3], [4, 5, 6]])
# array([[1, 2, 3],
# [4, 5, 6]])np.arange([start, ]stop, [step, ]dtype=None)
Retourne un ndarray avec des valeurs réparties régulièrement sur l’intervalle demandé avec un pas (step).
start : valeur de début (facultatif)stop : valeur de finstep : saut entre chaque valeur (par défaut 1)dtype : type choisiDe la même façon que pour la fonction range() de Python, la valeur stop ne fait pas partie des valeurs retournées.
np.arange(1, 5)
# array([1, 2, 3, 4])
np.arange(5.0)
# array([0., 1., 2., 3., 4.])
np.arange(1, 3, 0.5)
# array([1. , 1.5, 2. , 2.5])
np.arange(10)
# array([0, 1, 2, 3, 4, 5, 6, 7, 8, 9])np.linspace(start, stop, num=50, endpoint=True, retstep=False, dtype=None, axis=0)
Retourne un ndarray avec un nombre défini de valeurs réparties régulièrement sur l’intervalle demandé.
start : valeur de début (facultatif)stop : valeur de finnum : nombre de valeurs souhaitées (50 par défaut)endpoint : inclure ou pas la valeur stop (True par défaut)retstep : si True, retourne un tuple (sample, step) au lieu de sample uniquement (False par défaut)dtype : type choisinp.linspace(1, 10)
# array([ 1. , 1.18367347, 1.36734694,
# 1.55102041, 1.73469388, 1.91836735,
# 2.10204082, 2.28571429, 2.46938776,
# 2.65306122, 2.83673469, 3.02040816,
# 3.20408163, 3.3877551 , 3.57142857,
# 3.75510204, 3.93877551, 4.12244898,
# 4.30612245, 4.48979592, 4.67346939,
# 4.85714286, 5.04081633, 5.2244898 ,
# 5.40816327, 5.59183673, 5.7755102 ,
# 5.95918367, 6.14285714, 6.32653061,
# 6.51020408, 6.69387755, 6.87755102,
# 7.06122449, 7.24489796, 7.42857143,
# 7.6122449 , 7.79591837, 7.97959184,
# 8.16326531, 8.34693878, 8.53061224,
# 8.71428571, 8.89795918, 9.08163265,
# 9.26530612, 9.44897959, 9.63265306,
# 9.81632653, 10. ])
np.linspace(1, 10, 3)
# array([ 1. , 5.5, 10. ])
np.linspace(1, 10, 3, endpoint=False)
# array([1., 4., 7.])
np.linspace(1, 2, 5, retstep=True)
# (array([1. , 1.25, 1.5 , 1.75, 2. ]), 0.25)np.zeros(shape, dtype=float, order='C')
np.ones(shape, dtype=None, order='C')
np.full(shape, fill_value, dtype=None, order='C')
Retourne un ndarray de géométrie demandée, rempli avec des 0 ou 1 ou la valeur demandée.
shape : géométrie désiréefill_value : valeur d’initialisation pour chaque élément du ndarray.np.zeros(5, dtype=int)
# array([0, 0, 0, 0, 0])
np.zeros((2, 3), dtype=int)
# array([[0, 0, 0],
# [0, 0, 0]])
np.ones(5, dtype=int)
# array([1, 1, 1, 1, 1])
np.ones((2, 3), dtype=int)
# array([[1, 1, 1],
# [1, 1, 1]])
np.full(5, -1, dtype=int)
# array([-1, -1, -1, -1, -1])
np.full((2, 3), 5, dtype=int)
# array([[5, 5, 5],
# [5, 5, 5]])np.zeros_like(a, dtype=None, order='K', subok=True)
np.ones_like(a, dtype=None, order='K', subok=True)
np.full_like(a, fill_value, dtype=None, order='K', subok=True)
Retourne un ndarray de géométrie identique au ndarray passé, rempli avec des 0 ou 1 ou la valeur demandée.
a : objet ndarray dont on récupère la géométriefill_value : valeur d’initialisation pour chaque élément du ndarray.a = np.arange(21).reshape(3, 7)
a
# array([[ 0, 1, 2, 3, 4, 5, 6],
# [ 7, 8, 9, 10, 11, 12, 13],
# [14, 15, 16, 17, 18, 19, 20]])
np.zeros_like(a)
# array([[0, 0, 0, 0, 0, 0, 0],
# [0, 0, 0, 0, 0, 0, 0],
# [0, 0, 0, 0, 0, 0, 0]])
np.ones_like(a)
# array([[1, 1, 1, 1, 1, 1, 1],
# [1, 1, 1, 1, 1, 1, 1],
# [1, 1, 1, 1, 1, 1, 1]])
np.full_like(a, 5)
# array([[5, 5, 5, 5, 5, 5, 5],
# [5, 5, 5, 5, 5, 5, 5],
# [5, 5, 5, 5, 5, 5, 5]])np.random.rand(d0, d1, ..., dn)
Retourne un ndarray avec la géométrie définie par les arguments contenant des valeurs aléatoires de distribution uniforme [0, 1) (0 <= x < 1).
d0, d1, …, dn : les dimensions pour chaque axe.np.random.randn(d0, d1, ..., dn)
Retourne un ndarray avec la géométrie définie par les arguments contenant des valeurs aléatoires de distribution gaussienne (moyenne de 0 et variance de 1).
d0, d1, …, dn : les dimensions pour chaque axe.np.random.randint(low, high=None, size=None, dtype='l')
Retourne un ndarray avec la géométrie définie par size contenant des valeurs entières aléatoires de distribution discrete uniforme.
low : valeur inférieurehigh : valeur supérieure (non-inclue). Si elle n’est pas donnée, alors ce sera entre 0 et lowsize : entier donnant la dimension ou tuple décrivant la géométrie du ndarray retourné. Si il n’est pas donné, retournera un scalaire.dtype : le datatype désiré.np.random.random(size=None)
Retourne un ndarray avec la géométrie définie par size contenant des valeurs aléatoires de distribution uniforme [0, 1) (0 <= x < 1).
np.random.shuffle(x)
Modifie un ndarray en mélangeant ses éléments selon son premier axe.
x : ndarray à mélangernp.random.choice(a, size=None, replace=True, p=None)
Retourne un ndarray d’éléments choisis au hasard d’un 1-D array
a : 1-D array duquel seront pris les éléments choisis au hasard. Si c’est un entier, alors il est généré comme si c’était np.arange(a)size : géométrie du ndarray désirép : 1-D array décrivant la probabilité pour chaque élément. S’il n’est pas donné, ils sera fait avec une distribution uniforme parmi toutes les valeurs de aPlus de fonctions encore :  Manuel Scipy
Manuel Scipy
np.random.rand(2, 3)
# array([[0.77604531, 0.9128542 , 0.32148 ],
# [0.7614714 , 0.22650356, 0.06662943]])
np.random.randn(2, 3)
# array([[ 0.70331456, -0.45092623, 1.22391135],
# [-0.13710605, -0.26827598, 0.32249217]])
np.random.randint(1, 7, 10)
# array([2, 1, 3, 6, 1, 1, 5, 2, 1, 4])
np.random.randint(1, 300, 10000).min()
# 1
np.random.randint(1, 300, 10000).max()
# 299
np.random.random((3, 2))
# array([[0.32833677, 0.8962646 ],
# [0.05753724, 0.06057001],
# [0.76465039, 0.23744694]])
a = np.arange(20)
np.random.shuffle(a)
a
# array([ 2, 4, 5, 13, 7, 10, 6, 0, 16, 15, 17, 19,
# 12, 14, 18, 9, 3, 11, 1, 8])
np.random.choice(10, (2, 3))
# array([[0, 9, 2],
# [6, 1, 3]])
np.random.choice(['a', 'b', 'c'], (2, 3))
# array([['c', 'b', 'c'],
# ['b', 'a', 'c']], dtype='<U1')
np.random.choice(['a', 'b', 'c'], (2, 5), p=[0.2, 0.7, 0.1])
# array([['c', 'a', 'b', 'b', 'b'],
# ['b', 'b', 'c', 'b', 'a']], dtype='<U1')np.fromfunction(function, shape, kwargs)
Construit un ndarray initialisé avec les valeurs retournées par une fonction fn(x, y, z) pour l’élément à la position (x, y, z).
function : la fonction appelée avec les N paramètres représentant les N indexes de la matrice.
Pour un shape valant (3, 3), les 2 paramètres vaudront :
array([[0 0 0]
[1 1 1]
[2 2 2]])et
array([[0 1 2]
[0 1 2]
[0 1 2]])shape : la géométrie du ndarray demandé.
dtype : le datatype souhaité
np.fromfunction(lambda x, y: x * y, (5, 5))
# array([[ 0., 0., 0., 0., 0.],
# [ 0., 1., 2., 3., 4.],
# [ 0., 2., 4., 6., 8.],
# [ 0., 3., 6., 9., 12.],
# [ 0., 4., 8., 12., 16.]])
def init_my_array(x, y):
a = x + y
a[x < y] = 0
return a
np.fromfunction(init_my_array, (5, 5), dtype=int)
# array([[0, 0, 0, 0, 0],
# [1, 2, 0, 0, 0],
# [2, 3, 4, 0, 0],
# [3, 4, 5, 6, 0],
# [4, 5, 6, 7, 8]])numpy.meshgrid(xi, kwargs)
Construit N ndarray X1, X2, …, Xn.
x1, x2, x3, … : 1-D ndarray représentant les coordonnées de la grille.Ni = len(xi)indexing : parmi (xy, ij), défaut : xy
xy : indexation cartésienne
N2, N1, N3, N4, …)x1 sont réparties sur le 2ème axe, puis dupliquées sur les autres axes de X1.x2 sont réparties sur le 1er axe, puis dupliquées sur les autres axes de X2.x3 sont réparties sur le rème axe, puis dupliquées sur les autres axes de X3.ij : indexation matricielle
N1, N2, N3, N4, …)x1 sont réparties sur le 1er axe, puis dupliquées sur les autres axes de X1.x2 sont réparties sur le 2ème axe, puis dupliquées sur les autres axes de X2.x3 sont réparties sur le rème axe, puis dupliquées sur les autres axes de X3.Note : Cette méthode est très utile lorsqu’on souhaite évaluer une fonction sur toute une surface dans un plan, ou tout un espace (3-D ou plus).
# Exemple 2-D
x = np.arange(10)
y = np.arange(5)
xv, yv = np.meshgrid(x, y)
xv
# array([[0, 1, 2, 3, 4, 5, 6, 7, 8, 9],
# [0, 1, 2, 3, 4, 5, 6, 7, 8, 9],
# [0, 1, 2, 3, 4, 5, 6, 7, 8, 9],
# [0, 1, 2, 3, 4, 5, 6, 7, 8, 9],
# [0, 1, 2, 3, 4, 5, 6, 7, 8, 9]])
yv
# array([[0, 0, 0, 0, 0, 0, 0, 0, 0, 0],
# [1, 1, 1, 1, 1, 1, 1, 1, 1, 1],
# [2, 2, 2, 2, 2, 2, 2, 2, 2, 2],
# [3, 3, 3, 3, 3, 3, 3, 3, 3, 3],
# [4, 4, 4, 4, 4, 4, 4, 4, 4, 4]])
# Exemple 3-D
x = np.arange(2)
y = np.arange(2, 5)
z = np.arange(5, 9)
xv, yv, zv = np.meshgrid(x, y, z)
xv.shape
# (3, 2, 4)
xv
# array([[[0, 0, 0, 0],
# [1, 1, 1, 1]],
#
# [[0, 0, 0, 0],
# [1, 1, 1, 1]],
#
# [[0, 0, 0, 0],
# [1, 1, 1, 1]]])
xv[0,:,0]
# array([0, 1])
yv
# array([[[2, 2, 2, 2],
# [2, 2, 2, 2]],
#
# [[3, 3, 3, 3],
# [3, 3, 3, 3]],
#
# [[4, 4, 4, 4],
# [4, 4, 4, 4]]])
yv[:,0,0]
# array([2, 3, 4])
zv
# array([[[5, 6, 7, 8],
# [5, 6, 7, 8]],
#
# [[5, 6, 7, 8],
# [5, 6, 7, 8]],
#
# [[5, 6, 7, 8],
# [5, 6, 7, 8]]])
zv[0,0,:]
# array([5, 6, 7, 8])Similaire à np.meshgrid(..., indexing='ij')
Construit un ndarray condensé des N meshgrid
np.mgrid[]
Si l’élément de pas n’est pas complexe (ex. [1:5]), alors la valeur de fin n’est pas comprise.
Si l’élément de pas est complexe (ex. [1:5:17j]), alors il devient le nombre de valeurs demandées et la valeur de fin est comprise.
np.mgrid[1:2:5j, 0:3].shape
# (2, 5, 3)
X, Y = np.mgrid[1:2:5j, 0:3]
X
# array([[1. , 1. , 1. ],
# [1.25, 1.25, 1.25],
# [1.5 , 1.5 , 1.5 ],
# [1.75, 1.75, 1.75],
# [2. , 2. , 2. ]])
Y
# array([[0., 1., 2.],
# [0., 1., 2.],
# [0., 1., 2.],
# [0., 1., 2.],
# [0., 1., 2.]])Comme décrit plus bas, il est possible qu’on obtienne d’un appel à une fonction NumPy une vue sur un objet. Cela a pour conséquences que lorsqu’on modifie l’un, les modifications sont appliquées également à l’autre. Si ce comportement ne nous convient pas, il est possible demander une copie neuve dans un nouvel objet. np.copy() fait cela.
# Sans copie
a = np.arange(5)
a # array([0, 1, 2, 3, 4])
b = a[2:4]
b[0] = 0
a # array([0, 1, 0, 3, 4])
# Avec copie
a = np.arange(5)
a # array([0, 1, 2, 3, 4])
b = np.copy(a[2:4])
b[0] = 0
a # array([0, 1, 2, 3, 4])Propriété retournant le type des éléments du ndarray
a = np.array([1, 2, 3, 4])
a.dtype
# dtype('int64')Propriété retournant la géométrie de l’objet ndarray.
Il peut être assigné une valeur, dans quel cas, cela changera la géométrie de l’objet.
a = np.linspace(1, 6, 6)
a
Out[108]: array([1., 2., 3., 4., 5., 6.])
a.shape
# (6,)
a.shape = (3, 2)
a
# array([[1., 2.],
# [3., 4.],
# [5., 6.]])
a.shape = (3, 3)
# ValueError: cannot reshape array of size 6 into shape (3,3)numpy.reshape(a, newshape, order='C')
ndarray.reshape(newshape, order='C')
Permet de modifier la géométrie de l’objet ndarray. De la même façon que ndarray.shape décrit ci-dessus.
a : objet à manipulernewshape : entier ou tuple d’entiers décrivant la nouvelle géométrie désirée. S’il est donné un entier, alors cela retourne un ndarray à 1 dimension. Il est possible de donner -1 sur une dimension, dans ce cas, la valeur est calculée en fonction de la taille de l’objet.np.arange(12).reshape((3, 4))
# array([[ 0, 1, 2, 3],
# [ 4, 5, 6, 7],
# [ 8, 9, 10, 11]])
np.arange(12).reshape((4, -1))
# array([[ 0, 1, 2],
# [ 3, 4, 5],
# [ 6, 7, 8],
# [ 9, 10, 11]])
np.arange(12).reshape((2, 2, 3))
# array([[[ 0, 1, 2],
# [ 3, 4, 5]],
#
# [[ 6, 7, 8],
# [ 9, 10, 11]]])a = np.arange(12).reshape(3, 4)
a
# array([[ 0, 1, 2, 3],
# [ 4, 5, 6, 7],
# [ 8, 9, 10, 11]])
a.T
# array([[ 0, 4, 8],
# [ 1, 5, 9],
# [ 2, 6, 10],
# [ 3, 7, 11]])Il est possible d’accéder à un élément ou un ensemble d’éléments à l’intérieur d’un ndarray avec l’indexing et le slicing. Cela se fait en donnant le/les index entre [].
Lorsque la valeur est positive, on indexe depuis le début du ndarray, lorsqu’elle est négative, depuis la fin.
Pour un ndarray à multiple dimensions, on sépare chaque index (ou slice) avec une ,
Pour faire du slicing, on va donner 2 ou 3 valeurs, séparées d’un :. Cela sera [start:end] ou [start:end:step]
start : position de départ (commence par 0 depuis la gauche, et par -1 depuis la droite)end : position de fin (non-inclue)step : pas sur l’indexe (peut être négatif pour aller de droite à gauche)Cas particulier, on peut demander la totalité avec le simple caractère :
 Note importante : Lorsque l’on fait un slicing, on obtient une vue sur les données originales (ce qui limite la quantité de mémoire allouée).
Note importante : Lorsque l’on fait un slicing, on obtient une vue sur les données originales (ce qui limite la quantité de mémoire allouée).
a = np.arange(10)
a
# array([0, 1, 2, 3, 4, 5, 6, 7, 8, 9])
a[2]
# 2
a[-2]
# 8
a[1:4]
# array([1, 2, 3])
a[1:8:2]
# array([1, 3, 5, 7])
a[::-1]
# array([9, 8, 7, 6, 5, 4, 3, 2, 1, 0])
a = np.arange(10).reshape((2, -1))
a
# array([[0, 1, 2, 3, 4],
# [5, 6, 7, 8, 9]])
a[1] # ou a[1, :]
# array([5, 6, 7, 8, 9])
a[:, 1]
# array([1, 6])Il est possible de donner un tableau d’index. Cela retournera un ndarray de la même dimension avec les valeurs correspondantes.
a = np.arange(7)[::-1]
a
# array([6, 5, 4, 3, 2, 1, 0])
i = np.array([1, 5, 2, 2, 6])
a[i]
# array([5, 1, 4, 4, 0])
a = np.arange(50).reshape(5, 10)
a
# array([[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9],
# [10, 11, 12, 13, 14, 15, 16, 17, 18, 19],
# [20, 21, 22, 23, 24, 25, 26, 27, 28, 29],
# [30, 31, 32, 33, 34, 35, 36, 37, 38, 39],
# [40, 41, 42, 43, 44, 45, 46, 47, 48, 49]])
i = np.array([1, 2])
j = np.array([1, 4])
a[i, j]
# [11 24]Il est possible filtrer à l’aide d’un masque booléen un ndarray. Pour cela, il est nécessaire que le masque soit de la même dimension que l’objet filtré.
a = np.arange(8, 13)[::-1]
a
# array([12, 11, 10, 9, 8])
m = a>10
m
# array([ True, True, False, False, False])
a[m]
# array([12, 11])Pour chaque syntaxe où l’on obtient une vue à l’aide d’index ou d’un slicing, il est possible de l’utiliser dans une affectation, ce qui viendra écraser les valeurs correspondantes.
a = np.arange(10)
a[3:5] = 0
a
# array([0, 1, 2, 0, 0, 5, 6, 7, 8, 9])
a[4:7] = np.arange(3)[::-1]
a
# array([0, 1, 2, 0, 2, 1, 0, 7, 8, 9])
a[np.array([7, 5, 9])] -= 1
a
# array([0, 1, 2, 0, 2, 0, 0, 6, 8, 8])
a[a>5] = 0
a
# array([0, 1, 2, 0, 2, 0, 0, 0, 0, 0])L’ordre de lecture des axes d’un ndarray est le suivant :
[])[] avant le dernier axe[].Ainsi, pour un ndarray à 2 dimensions :
Pour un ndarray à 3 dimensions :
a = np.arange(24).reshape((2, 3, 4))
a
# array([[[ 0, 1, 2, 3],
# [ 4, 5, 6, 7],
# [ 8, 9, 10, 11]],
#
# [[12, 13, 14, 15],
# [16, 17, 18, 19],
# [20, 21, 22, 23]]])
a[0, 1, 2]
# 6
a[0, :, :]
# array([[ 0, 1, 2, 3],
# [ 4, 5, 6, 7],
# [ 8, 9, 10, 11]])
a[:, 0, :]
# array([[ 0, 1, 2, 3],
# [12, 13, 14, 15]])
a[:, :, 0]
# array([[ 0, 4, 8],
# [12, 16, 20]])
np.ravel(a, order='C')
a : l’objet array à transformerndarray.ravel([order])
Retourne un 1-D array contenant tous les éléments de l’array d’origine.
a = np.array([[1, 2, 3], [4, 5, 6]])
a.ravel()
# array([1, 2, 3, 4, 5, 6])
np.ravel(a)
# array([1, 2, 3, 4, 5, 6])np.resize(a, new_shape)
Retourne un nouvel ndarray avec la géométrie spécifiée.
Si la nouvelle taille est plus grande que l’originale, alors le nouvel ndarray est initialisé avec une répétition de la copie du contenu.
Si la nouvelle taille est plus petite, alors seuls les données qui passent dans la nouvelle taille sont copiées.
a : le array à transformernew_shape : la nouvelle géométrie demandée
ndarray.resize(new_shape, refcheck=True)
Change la géométrie de l’objet ndarray sur lui-même.
Si la nouvelle taille est plus grande que l’originale, alors le ndarray est complété avec des 0.
Si la nouvelle taille est plus petite, alors seuls les données qui passent dans la nouvelle taille sont conservés.
new_shape : la nouvelle géométrie demandéea = np.array([[1, 2, 3], [4, 5, 6]])
np.resize(a, (6, 2))
# array([[1, 2],
# [3, 4],
# [5, 6],
# [1, 2],
# [3, 4],
# [5, 6]])
np.resize(a, 3)
# array([1, 2, 3])
###
a = np.array([[1, 2, 3], [4, 5, 6]])
a.resize((6, 2))
a
# array([[1, 2],
# [3, 4],
# [5, 6],
# [0, 0],
# [0, 0],
# [0, 0]])
a = np.array([[1, 2, 3], [4, 5, 6]])
a.resize(3)
a
# array([1, 2, 3])np.repeat(a, repeats, axis=None)
Retourne un ndarray contenant une répétition des éléments d’un array
a : array à répéterrepeats : le nombre de répétitions désiré (entier ou tableau d’entiers)axis : l’axe selon lequel faire la répétitionnp.repeat(3, 4)
# array([3, 3, 3, 3])
a = np.arange(4).reshape(2, 2)
a
# array([[0, 1],
# [2, 3]])
np.repeat(a, 2)
# array([0, 0, 1, 1, 2, 2, 3, 3])
np.repeat(a, (2, 2), 0)
# array([[0, 1],
# [0, 1],
# [2, 3],
# [2, 3]])
np.repeat(a, (2, 2), 1)
# array([[0, 0, 1, 1],
# [2, 2, 3, 3]])numpy.tile(A, reps)
Construit un ndarray en répétant l’array donné un nombre de fois désiré
A : l’array à répéterreps : le nombre de répétitions à effectuer selon chaque axenp.tile(1, (2, 3))
# array([[1, 1, 1],
# [1, 1, 1]])
np.tile([1, 2, 3], (2, 3))
# array([[1, 2, 3, 1, 2, 3, 1, 2, 3],
# [1, 2, 3, 1, 2, 3, 1, 2, 3]])
a = np.arange(6).reshape(2, 3)
a
# array([[0, 1, 2],
# [3, 4, 5]])
np.tile(a, (1, 2))
# array([[0, 1, 2, 0, 1, 2],
# [3, 4, 5, 3, 4, 5]])
np.tile(a, (2, 1))
# array([[0, 1, 2],
# [3, 4, 5],
# [0, 1, 2],
# [3, 4, 5]])
np.tile(a, (2, 3, 4))
# array([[[0, 1, 2, 0, 1, 2, 0, 1, 2, 0, 1, 2],
# [3, 4, 5, 3, 4, 5, 3, 4, 5, 3, 4, 5],
# [0, 1, 2, 0, 1, 2, 0, 1, 2, 0, 1, 2],
# [3, 4, 5, 3, 4, 5, 3, 4, 5, 3, 4, 5],
# [0, 1, 2, 0, 1, 2, 0, 1, 2, 0, 1, 2],
# [3, 4, 5, 3, 4, 5, 3, 4, 5, 3, 4, 5]],
#
# [[0, 1, 2, 0, 1, 2, 0, 1, 2, 0, 1, 2],
# [3, 4, 5, 3, 4, 5, 3, 4, 5, 3, 4, 5],
# [0, 1, 2, 0, 1, 2, 0, 1, 2, 0, 1, 2],
# [3, 4, 5, 3, 4, 5, 3, 4, 5, 3, 4, 5],
# [0, 1, 2, 0, 1, 2, 0, 1, 2, 0, 1, 2],
# [3, 4, 5, 3, 4, 5, 3, 4, 5, 3, 4, 5]]])numpy.vstack(tup)
numpy.hstack(tup)
Empile des ndarrays verticalement/horizontalement
tup : séquence de ndarraysa = np.arange(5)
a
# array([0, 1, 2, 3, 4])
b = np.arange(10, 15)
b
# array([10, 11, 12, 13, 14])
np.vstack((a, b))
# array([[ 0, 1, 2, 3, 4],
# [10, 11, 12, 13, 14]])
np.hstack((a, b))
# array([ 0, 1, 2, 3, 4, 10, 11, 12, 13, 14])np.column_stack(tup)
Empile des 1-D arrays en colonnes dans un 2-D ndarray
tup : séquence de 1-D ou 2-D arrays à empiler. S’il s’agit d’un 2-D array, il est empilé tel quel.a = np.arange(5)
b = np.arange(10, 15)
c = np.arange(20, 25)
abc = np.column_stack((a, b, c))
abc
# array([[ 0, 10, 20],
# [ 1, 11, 21],
# [ 2, 12, 22],
# [ 3, 13, 23],
# [ 4, 14, 24]])
d = np.arange(30, 35)
np.column_stack((abc, d))
# array([[ 0, 10, 20, 30],
# [ 1, 11, 21, 31],
# [ 2, 12, 22, 32],
# [ 3, 13, 23, 33],
# [ 4, 14, 24, 34]])np.split(ary, indices_or_sections, axis=0)
Découpe un ndarray en multiple sous-ndarrays selon l’axe choisi.
ary : le ndaray à découperindices_or_sections : entier ou 1-D array décrivant la/les positions de découpe.axis : donne l’axe selon lequel la découpe doit être faite.a = np.arange(24).reshape(2, 3, 4)
a
array([[[ 0, 1, 2, 3],
[ 4, 5, 6, 7],
[ 8, 9, 10, 11]],
[[12, 13, 14, 15],
[16, 17, 18, 19],
[20, 21, 22, 23]]])
np.split(a, 2, 0)
# [array([[[ 0, 1, 2, 3],
# [ 4, 5, 6, 7],
# [ 8, 9, 10, 11]]]),
# array([[[12, 13, 14, 15],
# [16, 17, 18, 19],
# [20, 21, 22, 23]]])]
np.split(a, 3, 1)
# [array([[[ 0, 1, 2, 3]],
#
# [[12, 13, 14, 15]]]),
#
# array([[[ 4, 5, 6, 7]],
#
# [[16, 17, 18, 19]]]),
#
# array([[[ 8, 9, 10, 11]],
#
# [[20, 21, 22, 23]]])]
np.split(a, [2,], 1)
# [array([[[ 0, 1, 2, 3],
# [ 4, 5, 6, 7]],
#
# [[12, 13, 14, 15],
# [16, 17, 18, 19]]]),
#
# array([[[ 8, 9, 10, 11]],
#
# [[20, 21, 22, 23]]])]
np.split(a, 4, 2)
# [array([[[ 0],
# [ 4],
# [ 8]],
#
# [[12],
# [16],
# [20]]]),
#
# array([[[ 1],
# [ 5],
# [ 9]],
#
# [[13],
# [17],
# [21]]]),
#
# array([[[ 2],
# [ 6],
# [10]],
#
# [[14],
# [18],
# [22]]]),
#
# array([[[ 3],
# [ 7],
# [11]],
#
# [[15],
# [19],
# [23]]])]
np.split(a, 2, 2)
# [array([[[ 0, 1],
# [ 4, 5],
# [ 8, 9]],
#
# [[12, 13],
# [16, 17],
# [20, 21]]]),
#
# array([[[ 2, 3],
# [ 6, 7],
# [10, 11]],
#
# [[14, 15],
# [18, 19],
# [22, 23]]])]np.all(a, axis=None, out=None, keepdims=<no value>)
Teste si tous les éléments selon un axe donné sont évalués à vrai (True) et retourne un scalaire (si aucun axe n’est fourni) ou un ndarray contenant la réponse pour chaque axe.
a : array parcouruaxis : axe choisi (si aucun axe n’est donné alors le résultat concerne toute le ndarray)np.all([[True, True], [False, True]])
# False
np.all([[True, True], [False, True]], 0)
# array([False, True])
np.all([[True, True], [False, True]], 1)
# array([ True, False])
a = np.linspace(-4, 4, 9, dtype=int).reshape(3, 3)
a
# array([[-4, -3, -2],
# [-1, 0, 1],
# [ 2, 3, 4]])
np.all(a, 0)
# array([ True, False, True])Le terme de broadcasting décrit la façon dont numpy adapte deux objets scalaires ou ndarrays de géométrie différentes dans le but d’effectuer une opération entre elles.
Chaque axe est comparé, allant du dernier au premier. Celui de plus petite dimension est broadcasté jusqu’à obtenir la même dimension que le plus grand. Ainsi nous obtenons 2 objets de géométrie identique.
Deux dimensions sont compatibles si :
1Si les dimensions ne sont pas compatibles, une exception ValueError est lancée.
Exemples de broadcast qui fonctionnent :
A (2d array): 5 x 4
B (1d array): 1
Result (2d array): 5 x 4A (4d array): 8 x 1 x 6 x 1
B (3d array): 7 x 1 x 5
Result (4d array): 8 x 7 x 6 x 5Exemples de broadcast qui ne peuvent pas fonctionner :
A (2d array): 5 x 4
B (1d array): 2A (4d array): 8 x 1 x 6 x 1
B (3d array): 8 x 1 x 2 x 1np.array([1, 2]) + np.array([[10], [20]])
# array([[11, 12],
# [21, 22]])
np.arange(10) * 3
# array([ 0, 3, 6, 9, 12, 15, 18, 21, 24, 27])
a = np.arange(27).reshape(3, 3, 3)
a * np.arange(3)
# array([[[ 0, 1, 4],
# [ 0, 4, 10],
# [ 0, 7, 16]],
#
# [[ 0, 10, 22],
# [ 0, 13, 28],
# [ 0, 16, 34]],
#
# [[ 0, 19, 40],
# [ 0, 22, 46],
# [ 0, 25, 52]]])
a * np.arange(3)[:, np.newaxis]
# array([[[ 0, 0, 0],
# [ 3, 4, 5],
# [12, 14, 16]],
#
# [[ 0, 0, 0],
# [12, 13, 14],
# [30, 32, 34]],
#
# [[ 0, 0, 0],
# [21, 22, 23],
# [48, 50, 52]]])
a * np.arange(3)[:, np.newaxis, np.newaxis]
# array([[[ 0, 0, 0],
# [ 0, 0, 0],
# [ 0, 0, 0]],
#
# [[ 9, 10, 11],
# [12, 13, 14],
# [15, 16, 17]],
#
# [[36, 38, 40],
# [42, 44, 46],
# [48, 50, 52]]])
a * np.arange(4)[:, np.newaxis]
# ----------------------------------------------------
# ValueError Traceback (most recent call last)
# <ipython-input-107-1868fd69169b> in <module>
# ----> 1 np.arange(27).reshape(3, 3, 3) *
# np.arange(4)[:, np.newaxis]
#
# ValueError: operands could not be broadcast together
# with shapes (3,3,3) (4,1)Ces opérations sont effectuées, après un broadcast automatique, éléments par éléments (contrairement au sens mathématique de ces opérations).
Voici 2 exemples pour la multiplication a * b, selon la géométrie initiale :
\[ \begin{bmatrix} a_{0 0} \\ a_{1 0} \\ a_{2 0} \end{bmatrix} * \begin{bmatrix} b_{0 0} & b_{0 1} & b_{0 2} \\ b_{1 0} & b_{1 1} & b_{1 2} \\ b_{2 0} & b_{2 1} & b_{2 2} \end{bmatrix} = \begin{bmatrix} a_{0 0} * b_{0 0} & a_{0 0} * b_{0 1} & a_{0 0} * b_{0 2} \\ a_{1 0} * b_{1 0} & a_{1 0} * b_{1 1} & a_{1 0} * b_{1 2} \\ a_{2 0} * b_{2 0} & a_{2 0} * b_{2 1} & a_{2 0} * b_{2 2} \end{bmatrix} \]
\[ \begin{bmatrix} a_{0} & a_{1} & a_{2} \end{bmatrix} * \begin{bmatrix} b_{0 0} & b_{0 1} & b_{0 2} \\ b_{1 0} & b_{1 1} & b_{1 2} \\ b_{2 0} & b_{2 1} & b_{2 2} \end{bmatrix} = \begin{bmatrix} a_{0} * b_{0 0} & a_{1} * b_{0 1} & a_{2} * b_{0 2} \\ a_{0} * b_{1 0} & a_{1} * b_{1 1} & a_{2} * b_{1 2} \\ a_{0} * b_{2 0} & a_{1} * b_{2 1} & a_{2} * b_{2 2} \end{bmatrix} \]
np.add(x1, x2, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])équivalent à :
x1 + x2
np.subtract(x1, x2, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])équivalent à :
x1 - x2
np.multiply(x1, x2, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])équivalent à :
x1 * x2
np.divide(x1, x2, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])équivalent à :
x1 / x2
np.power(x1, x2, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])équivalent à :
x1 ** x2
x1 et x2 : scalaires ou arrays de valeurs sur lesquelles nous souhaitons calculer l’opération.a = a = np.arange(3)[:, np.newaxis]
a
# array([[0],
# [1],
# [2]])
b = np.arange(9).reshape(3, 3)
b
# array([[0, 1, 2],
# [3, 4, 5],
# [6, 7, 8]])
a * b
# array([[ 0, 0, 0],
# [ 3, 4, 5],
# [12, 14, 16]])
b / 10
# array([[0. , 0.1, 0.2],
# [0.3, 0.4, 0.5],
# [0.6, 0.7, 0.8]])
b ** 3
# array([[ 0, 1, 8],
# [ 27, 64, 125],
# [216, 343, 512]])numpy.matmul(x1, x2, /, out=None, *,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])équivalent à :
x1 @ x2
Retourne un ndarray contenant le produit matriciel au sens algèbre linéaire.
x1 et x2 : arrays à multipliera = np.array([[1, 0],
[2, -1]])
b = np.array([[ 3, 4],
[-2, -3]])
a @ b
# array([[ 3, 4],
# [ 8, 11]])numpy.negative(x, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])équivalent à :
-x
Retourne un ndarray contenant tous les éléments de l’array fourni en inversant le signe.
x : arraya = np.arange(-4,4)
a
# array([-4, -3, -2, -1, 0, 1, 2, 3])
np.negative(a)
# array([ 4, 3, 2, 1, 0, -1, -2, -3])
-a
# array([ 4, 3, 2, 1, 0, -1, -2, -3])numpy.sum(a, axis=None, dtype=None, out=None,
keepdims=<no value>, initial=<no value>,
where=<no value>)ndarray.sum(axis=None, dtype=None, out=None,
keepdims=False, initial=0, where=True)Retourne un ndarray contenant la somme des éléments sur l’axe donné.
a : array sur lequel travailleraxis : axe selon lequel la somme sera effectuéeinitial : valeur de départ pour les sommesNOTE
a = np.arange(12).reshape(3, 4)
a
# array([[ 0, 1, 2, 3],
# [ 4, 5, 6, 7],
# [ 8, 9, 10, 11]])
a.sum(0)
# array([12, 15, 18, 21])
a.sum(1)
# array([ 6, 22, 38])
np.sum(np.arange(12))
# 66np.cumsum(a, axis=None, dtype=None, out=None)
ndarray.cumsum(axis=None, dtype=None, out=None)
Retourne un ndarray contenant la somme cumulative selon un axe donné.
a : array sur lequel travailleraxis : axe selon lequel la somme sera effectuéea = np.arange(12).reshape(3, 4)
a
# array([[ 0, 1, 2, 3],
# [ 4, 5, 6, 7],
# [ 8, 9, 10, 11]])
a.cumsum(0)
# array([[ 0, 1, 2, 3],
# [ 4, 6, 8, 10],
# [12, 15, 18, 21]])
a.cumsum(1)
# array([[ 0, 1, 3, 6],
# [ 4, 9, 15, 22],
# [ 8, 17, 27, 38]])np.amin(a, axis=None, out=None, keepdims=<no value>,
initial=<no value>, where=<no value>)ndarray.min(axis=None, out=None, keepdims=False,
initial=<no value>, where=True)np.amax(a, axis=None, out=None, keepdims=<no value>,
initial=<no value>, where=<no value>)ndarray.max(axis=None, out=None, keepdims=False,
initial=<no value>, where=True)Retourne un scalaire ou un ndarray donnant le minimum / maximum d’un array selon un axe donné.
a : array sur lequel travailleraxis : axe selon lequel la comparaison sera effectuée. Si aucun axe n’est donné, alors la recherche se fait sur l’objet en entier et un scalaire est retourné.a = np.arange(12).reshape(3, 4)
a
array([[ 0, 1, 2, 3],
[ 4, 5, 6, 7],
[ 8, 9, 10, 11]])
np.amin(a, 0)
# array([0, 1, 2, 3])
np.amin(a, 1)
# array([0, 4, 8])
np.amax(a, 0)
# array([ 8, 9, 10, 11])
np.amax(a, 1)
# array([ 3, 7, 11])
np.amin(a)
# 0
np.amax(a)
# 11np.argmin(a, axis=None, out=None) ndarray.argmin(axis=None, out=None)
np.argmax(a, axis=None, out=None) ndarray.argmax(axis=None, out=None)
Retourne un ndarray contenant les indices des valeurs minimum / maximum de l’array selon l’axe donné.
a : array sur lequel travailleraxis : axe selon lequel la comparaison sera effectuéea = np.random.choice(10, (3, 4))
a
# array([[7, 2, 7, 6],
# [8, 6, 6, 0],
# [7, 9, 1, 9]])
np.argmin(a, 0)
# array([0, 0, 2, 1])
np.argmin(a, 1)
# array([1, 3, 2])
np.argmax(a, 0)
# array([1, 2, 0, 2])
np.argmax(a, 1)
# array([0, 0, 1])np.mean(a, axis=None, dtype=None, out=None, keepdims=<no value>)
Retourne la valeur moyenne arithmétique (globale ou selon les axes spécifiés).
np.nanmean(a, axis=None, dtype=None, out=None, keepdims=<no value>)
Identique, mais ignore les valeurs NaN
np.var(a, axis=None, dtype=None, out=None, keepdims=<no value>)
Retourne la variance (globale ou selon les axes spécifiés).
np.nanvar(a, axis=None, dtype=None, out=None, keepdims=<no value>)
Identique, mais ignore les valeurs NaN
np.std(a, axis=None, dtype=None, out=None, keepdims=<no value>)
Retourne la déviation standard (globale ou selon les axes spécifiés).
np.nanstd(a, axis=None, dtype=None, out=None, keepdims=<no value>)
Identique, mais ignore les valeurs NaN
a : array sur lequel travailleraxis : None ou int ou tuple de inta = np.random.rand(20) * 50 + 10
np.mean(a), np.var(a), np.std(a)
# (39.43834962257571, 155.85423018128023, 12.484159169975374)
# With a NaN value
a[5] = np.NaN
np.mean(a), np.var(a), np.std(a)
# (nan, nan, nan)
np.nanmean(a), np.nanvar(a), np.nanstd(a)
# (38.8586547309587, 157.3361610540105, 12.543371199721808)np.sin(x, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])np.cos(x, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])np.tan(x, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])np.arcsin(x, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])np.arccos(x, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])np.arctan(x, /, out=None, *, where=True,
casting='same_kind', order='K',
dtype=None,
subok=True[, signature, extobj])Retourne un array de même type que fourni en entrée avec les valeurs calculées selon la fonction appelée.
x : scalaire ou array de valeurs sur lesquelles nous souhaitons calculer la fonction mathématiquenp.sin(np.pi/2)
# 1.0
np.cos(0)
# 1.0
np.arctan([0, 1])
# array([0. , 0.78539816])
np.sin(np.linspace(-np.pi, np.pi, 10))
# array([-1.22464680e-16, -6.42787610e-01,
# -9.84807753e-01, -8.66025404e-01,
# -3.42020143e-01, 3.42020143e-01,
# 8.66025404e-01, 9.84807753e-01,
# 6.42787610e-01, 1.22464680e-16])Les opérations de comparaison sont disponibles pour les objets ndarray. Elles retourne un ndarray comprenant le résultat de la comparaison, élément par élément.
x < y
x <= y
x > y
x >= y
x == y
x != y
x et y : objets ndarray
a = np.arange(-2,2)
a
# array([-2, -1, 0, 1])
b = -a
b
# array([ 2, 1, 0, -1])
a < b
# array([ True, True, False, False])
a == b
# array([False, False, True, False])np.pi
# 3.141592653589793np.e
# 2.718281828459045np.euler_gamma
# 0.5772156649015329np.inf
# inf
np.array([1]) / 0
# array([inf])np.NINF
# -inf
np.array([-1]) / 0
# array([-inf])np.NZERO
# -0.0
np.array([1]) / np.NZERO
# array([-inf])np.nan
nan pour “Not A Number”.
Couramment utilisé lorsque dans une série, il manque une mesure par exemple.
np.nan
# nan
np.log(-1)
# nannp.newaxis is None
# True
a = np.arange(0,5)
a
# array([0, 1, 2, 3, 4])
a[np.newaxis, :]
# array([[0, 1, 2, 3, 4]])
a[:, np.newaxis]
# array([[0],
# [1],
# [2],
# [3],
# [4]])
a[:, np.newaxis] * a
# array([[ 0, 0, 0, 0, 0],
# [ 0, 1, 2, 3, 4],
# [ 0, 2, 4, 6, 8],
# [ 0, 3, 6, 9, 12],
# [ 0, 4, 8, 12, 16]])Le language Python permet de créer et utiliser des tableaux nativement. La builtin list permet de créer un objet à 1 dimension, extensible à volonté et dont le typage est dynamique. La syntaxe raccourcie de list est simplement les crochets [ ].
L’objet list supporte quantités d’opérations qui sont décrites ici :  https://docs.python.org/3/tutorial/datastructures.html
https://docs.python.org/3/tutorial/datastructures.html
list([1, 2, 3])
# [1, 2, 3]
a_list = [1, 2, 3]
a_list[0]
# 1
a_list.append('je suis un texte')
a_list.extend([10, 11, 12])
a_list
# [1, 2, 3, 'je suis un texte', 10, 11, 12]Pour créer des tableaux à 2 ou N dimensions, il faudra alors créer des list de list (de list …)
a_matrix = [[1, 2, 3], [4, 5, 6], [7, 8, 9]]
a_matrix
# [[1, 2, 3], [4, 5, 6], [7, 8, 9]]
a_matrix.append([10, 11, 12])
a_matrix
# [[1, 2, 3], [4, 5, 6], [7, 8, 9], [10, 11, 12]]
a_cube = [[[ 1, 2, 3], [ 4, 5, 6], [ 7, 8, 9]],
[[10, 11, 12], [13, 14, 15], [16, 17, 18]],
[[19, 20, 21], [22, 23, 24], [25, 26, 27]]]list avec les objets Numpy de type np.arrayPython list |
Numpy np.array |
|---|---|
| natif au langage | requiert l’installation et l’import de numpy |
| typage dynamique | typage fixé à la création lors de la création de l’objet |
| peut être redimensionné | de taille fixe |
| plus gourmand en mémoire | moins d’espace en mémoire nécessaire |
| dédiée au stockage d’informations | dédié au travail sur des matrices |
| moins performant | plus performant en temps de calcul |
On voit bien ici les possibilités natives du langage Python. Elles sont plus axées vers des fonctionnalités de gestion et stockage d’information, plutôt que du travail sur des matrices comme nous en avons besoin dans du calcul scientifique.
Nous pouvons représenter l’espace mémoire d’une liste en Python de la façon suivante.
Les espaces mémoire sont représentés ici avec leur adresses et la valeur stockée.
Nous voyons que cela commence par un espace qui sert à décrire l’objet liste. Ensuite chaque élément de la liste connaît l’adresse où est stockée l’information cherchée. Il faudra donc faire un 2ème accès mémoire pour obtenir la valeur.
Ceci est le coût à payer du choix de Python de ne pas forcer le typage des données à l’intérieur d’une liste.
my_list = [5.75, 38.6, 1.94]
# Memory | Pointer to Data
# Address | Location
# --------|----------------
# ... | List
# ... | Meta
# ... | Information
# 0050h | 1204h
# 0054h | F1A3h
# 0058h | 2B87h
#
# 1204h | Meta
# ... | Information
# ... | 5.75
#
# 2B87h | Meta
# ... | Information
# ... | 1.94
#
# F1A3h | Meta
# ... | Information
# ... | 38.6Chaque Array Numpy peut être représenté en mémoire de la façon suivante.
À nouveau, nous avons un espace de méta-informations qui décrivent l’objet Array. Dans cet espace se trouve, entre autres, l’information du type de données stockées. Ainsi il est possible de stocker toutes les valeurs de façon contigüe.
my_ndarray = np.array([5.75, 38.6, 1.94])
# Memory | Pointer to Data
# Address | Location
# --------|----------------
# ... | Numpy
# ... | Array
# ... | Meta
# ... | Information
# 0050h | 5.75
# 0054h | 38.6
# 0058h | 1.94L’exercice consiste simplement à multiplier tous les éléments d’un tableau de valeurs par 3
Nous allons utiliser la librairie timeit pour chronométrer l’exécution d’une opération.
Pour ce faire, nous lui passons 3 arguments :
stmt (statement) : l’opération à chronométrersetup : les opérations à exécuter pour préparer le contexte nécessaire au stmtnumber : le nombre d’exécutions désiré (ce qui permet d’avoir une mesure moyenne)La fonction py_mult_by_3 va parcourir tous les éléments de la liste et multiplier chaque valeur par 3 pour les restocker à la même place.
import timeit
nb_run = 10000
nb_elems = 1000
my_list = list(range(nb_elems))
def py_mult_by_3(my_list):
for i in range(len(my_list)):
my_list[i] *= 3
return my_list
chrono = timeit.timeit(
"py_mult_by_3(my_list)",
setup="from _main_ import py_mult_by_3, my_list",
number=nb_run
)
print(chrono) # 2.4857738299997436Comme pour ci-dessus, nous utilisont timeit. Cette fois-ci, le stmt est le simple appel à la multiplication élément par élément.
import numpy as np
import timeit
nb_run = 10000
nb_elems = 1000
my_array = np.arange(nb_elems)
chrono = timeit.timeit(
"my_array *= 3",
setup="from _main_ import my_array",
number=nb_run
)
print(chrono) # 0.00918828900103108Ainsi l’on obtient les chronos de 2.49s avec l’utilisation des listes Python et de 0.00919s avec l’utilisation du numpy Array. Cela fait un gain de performance de 271x plus rapide!
En collaboration avec Thomas Lochmatter, un comparatif entre langages a été réalisé. Il s’agissait de coder l’agorithme du  crible d’Ératosthène (Sieve of Eratosthenes) dans différents langages et pour certains avec l’utilisation de librairies permettant une optimisation et d’en mesurer les performances (temps de compilation, exécution et impact mémoire).
crible d’Ératosthène (Sieve of Eratosthenes) dans différents langages et pour certains avec l’utilisation de librairies permettant une optimisation et d’en mesurer les performances (temps de compilation, exécution et impact mémoire).
Les codes en question sont décrits et disponibles ici :  https://viereck.ch/c/sieve/.
https://viereck.ch/c/sieve/.
Nous avons ensuite graphé le temps d’exécution que prend chaque solution en fonction du nombre de runs de ceux-ci. Voici ce que cela donne :
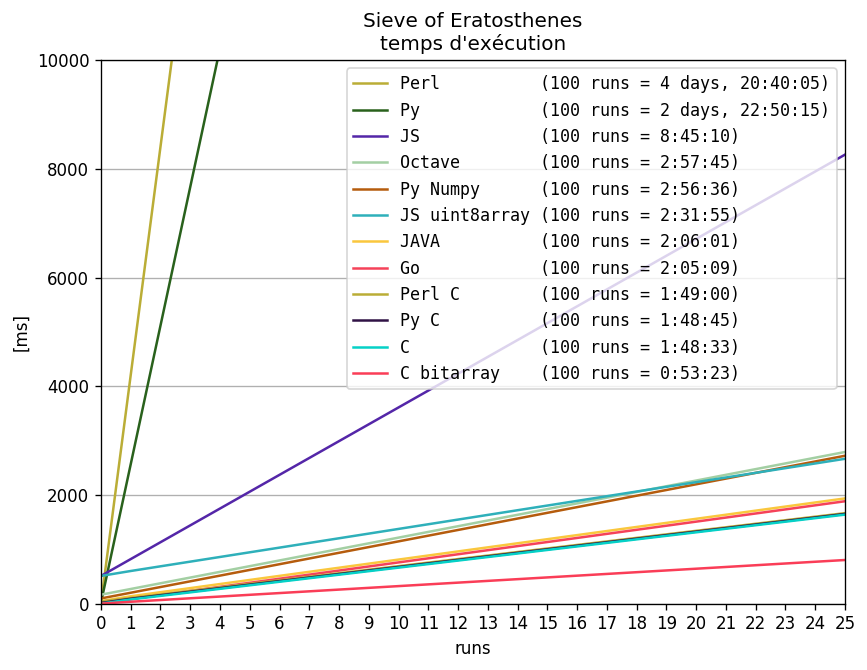
Une lecture rapide de ce graph, nous permet de placer les solutions testées dans 3 catégories.
En s’attardant sur les premiers de classement, on voit clairement C en tête, suivi directement par Python + C et Perl + C qui ont justement délégué le travail à optimiser à C. Viennent ensuite Go, JAVA, JS uint8array, Python + Numpy et Octave.
On voit bien là l’avantage qu’apporte une librairie tel que Numpy à Python!
Comme décrit en préambule, il faudra pondérer ces résultats avec le temps pour réaliser le développement, sa complexité, sa lisibilité, son extensibilité.
 Samuel Bancal, EPFL, ENAC-IT (2019-2020)
Samuel Bancal, EPFL, ENAC-IT (2019-2020)